Le vaccin fabriqué par les sociétés Biontech/Pfizer , nommé BNT162B, présente la particularité de ne pas être constitué d’un agent infectieux atténué ou inactivé classique mais d’une chimère de son matériel génétique modifié en laboratoire : il contient un ARN messager capable d’être traduit en une protéine « Spike » semblable à celle que l’on retrouve à la surface du virus.
National Institute of Allergy and Infectious Diseases (NIAID)
 Capture d’écran du logiciel Geniegen2 montrant la structure tridimensionnelle de la protéine Spike du virus
Capture d’écran du logiciel Geniegen2 montrant la structure tridimensionnelle de la protéine Spike du virus
Le code source de l’ARN du vaccin a été publié et est disponible sur le site de l’OMS (https://mednet-communities.net/inn/db/media/docs/11889.doc).
On trouve dans la banque de séquences de Geniegen2 des séquences nucléotidiques relatives au virus Sars-CoV-2. On dispose :
-
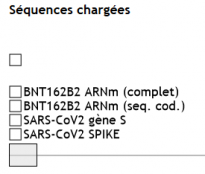 De la séquence complète de l’ARNm du vaccin BNT162B ;
De la séquence complète de l’ARNm du vaccin BNT162B ; - De la séquence codante de l’ARNm du vaccin ;
- De la séquence ARNm du gène S du virus codant pour la protéine Spike (S) ;
- De la séquence peptidique de la protéine spike présente à la surface du virus.
Les deux premières séquences n’ont pas la même longueur. La séquence complète présente dans le vaccin est constituée de 4282 nucléotides, contre 3822 pour la séquence codante.
Quelles sont les différences entre les deux séquences proposées ?
En utilisant l’outil d’alignement de Geniegen2 (Actions > Aligner les séquences sélectionnées), on obtient le résultat suivant :



On constate que la séquence complète du vaccin présente en amont de la séquence codante 54 nucléotides en plus, et 460 nucléotides de plus en aval de la séquence dont une succession d’adénines à partir du 4175e nucléotide.
Quels sont les rôles de ces parties supplémentaires ?
Le schéma de l’ARNm présenté sur l’article de l’OMS donne des explications.

La partie codante de l’ARNm (S protein_mut) est précédée :
- D’une coiffe (cap)
- D’une région 5’ non-traduite (5’ -UTR pour Untranslated Transcribed Region)
- D’une région codant pour un peptide signal de la glycoprotéine S (sig) A la suite de la partie codante, on trouve :
- Une région 3’ non traduite (3’ -UTR)
- Une queue polyA (poly(A)).
1 – La coiffe (1-2) (les positions des nucléotides sont données dans le document de l’OMS mais il est possible d’en déterminer certaines grâce à Geniegen2).
Le début de la séquence est une coiffe (« cap »). Cette coiffe a plusieurs fonctions, dont celle de montrer à la cellule l’origine de l’ARNm (ici une origine nucléaire). : cela évite à la cellule de détruire cet ARNm injecté lors de la vaccination. Les deux premiers nucléotides de la séquence (GA) jouent le rôle de coiffe.
2 – la région 5’ non traduite (3 - 54)
Pour réaliser une traduction la molécule d’ARNm est lue par les ribosomes dans le sens 5’-3’. Les régions 5’-UTR jouent un rôle fondamental dans la stabilité de la molécule d’ARNm et dans l’efficacité de la traduction.
Pour le vaccin, le 5’ UTR utilisé provient du gène de l’alpha globine. Ce gène est connu pour produire beaucoup de protéines de manière fiable. D’après le document de l’OMS, il ne s’agit pas tout à fait de la séquence UTR de l’alpha globine : c’est une version améliorée.
3 – Le peptide signal S (55-102)
Le début de la séquence codante code pour un peptide signal servant à l’adressage de la protéine après sa traduction. La localisation d’une protéine dans la cellule est essentielle à son bon fonctionnement. Or le lieu de production d’une protéine est souvent différent de son lieu d’action. L’adressage est l’ensemble des mécanismes qui permettent à une protéine d’être dirigée vers la bonne position.
4 - Partie codante
La partie codante de l’ARNm est en réalité constituée de 3777 nucléotides.
5- 3’-UTR (3880-4174)
Selon le document de l’OMS, la 3’-UTR du vaccin BioNTech / Pfizer a été choisie à partir de « l’amplificateur amino-terminal de l’ARNm scindé (AES) et de l’ARN ribosomal 12S encodé pour les mitochondries, pour conférer une stabilité à l’ARN et une expression protéique totale élevée ».
6- La queue polyA (4175-4284)
La fin de l’ARNm est polyadénylée : elle se termine par une centaine de nucléotides A (adénine) entrecoupés par un ’linker’ de 10 nucléotides : cette queue polyA est indispensable à la maturation et à l’activité du RNA messager qui la porte. Elle sera lentement digérée par les exonucléases du cytoplasme lorsque le messager sera actif. Lorsqu’elle sera trop réduite le messager vieilli sera détruit totalement.


En comparant avec GenieGen2 les séquences codantes de l’ARNm du vaccin et de l’ARNm du gène S codant pour les protéines spike, on observe des différences de nucléotides, plutôt localisées en 3e position de chaque codon.
Quelles conséquences ces différences dans la séquence peuvent-elles avoir sur la protéine néo-synthétisée ?
Pour le mettre en évidence, on peut alors souhaiter comparer la protéine spike du virus avec la protéine spike issue de la traduction de l‘ARNm du vaccin.
Avec GenieGen2, on traduit la séquence codante (Clic droit sur la séquence > Traduire en protéine). On peut alors comparer les deux séquences peptidiques. Les deux séquences sont comparables à 99,84%, ce qui représente deux acides aminés différents :

Dans le détail, l’ARNm du vaccin possède deux codons STOP (UGA) alors que l’ARNm naturel n’en contient qu’un (UAA).
Mais dans ce cas, pourquoi les chercheurs ont-ils changé deux acides aminés ?
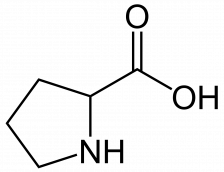 Ces deux acides aminés sont deux prolines.
Ces deux acides aminés sont deux prolines.
Les spicules fabriquées à partir de l’ARNm du vaccin ne seront pas portées par un virus. Or, il se trouve qu’une protéine spike non modifiée et non fixée sur un virus voit sa structure tridimensionnelle modifiée (elle se recroqueville). L’immunité acquise par ce vaccin n’agirait donc pas sur les protéines ’dressées’ du virus… En 2017, une équipe de chercheurs [1] a démontré que la double substitution de prolines dans en endroit précis permet aux protéines SARS-CoV-1 et MERS S de reprendre leur configuration ’dressée’, grâce à la rigidité de la proline.
Il reste une question importante : dans le code de la séquence du vaccin publié par l’OMS, on observe des nucléotides inattendus : Ψ. A quoi correspond cette notation ?
Lorsqu’on réalise par exemple un alignement des séquences du vaccin BNT162B2 complètes et codantes dans GenieGen 2, on observe que la séquence complète du vaccin est modifiée : les Ψ sont remplacés par des U (Uracile).

Le système immunitaire est très actif contre les virus : les ARNm d’origine virale sont normalement détruits rapidement. Or, il a été découvert [2] que si l’uracile de l’ARNm est remplacé par une molécule proche, le système immunitaire est trompé.
Donc dans le vaccin BioNTech / Pfizer, chaque U a été remplacé par du 1-méthyl-3’-pseudouridylyle, noté Ψ : cette substitution permet donc de ne pas alerter notre système immunitaire, le Ψ est accepté comme un U normal par les mécanismes cellulaires !
La comparaison des séquences publiées par l’OMS et disponibles dans la banque de séquences du logiciel Geniegen2 proposé par P. Cosentino permet donc de comprendre comment les chercheurs ont utilisé des connaissances issues de domaines variés pour construire une chimère génétique dont la traduction aboutit à la synthèse d’une protéine ressemblant à la protéine spike présente à la surface du virus. L’injection de ce vaccin vise à créer une mémoire immunitaire dirigée contre la protéine Spike du virus.
Place dans les programmes : Enseignement de spécialité en première
La Terre, la vie et l’organisation du vivant
Transmission, variation et expression du patrimoine génétique
L’expression du patrimoine génétique
Le code génétique est un système de correspondance, universel à l’ensemble du monde vivant, qui permet la traduction de l’ARN messager en protéines. L’information portée par une molécule d’ARN messager (le message génétique) est ainsi convertie en une information fonctionnelle (la séquence des acides aminés de la protéine).
Exemple de questions à envisager :
Ces informations pourraient constituer un document à destination des élèves, en lien avec l’utilisation de Genigen2.
On pourrait par exemple demander aux élèves de rechercher les astuces trouvées par les chercheurs pour :
1- faire croire aux cellules que l’ARNm n’est pas exogène afin d’éviter sa destruction par le système immunitaire.
2- faire exprimer une protéine Spike comparable à celle du virus afin de déclencher une réponse immunitaire efficace en cas de rencontre du virus.
Compétences travaillées
L’utilisation conjointe de Geniegen2 permet de travailler des compétences numériques. Par exemple, on peut citer :
- Recenser, extraire et exploiter des informations pour déterminer la composition d’un vaccin et son mode d’emploi ;
- Recenser, extraire et exploiter des informations pour déterminer les propriétés du code génétique (code génétique redondant, non ambigu,…).
- Utiliser des logiciels d’acquisition, de simulation et de traitement des données.
J.O. BOUDIER - Lycée Ch. de Gaulle - VANNES

